一、BLAST+下载
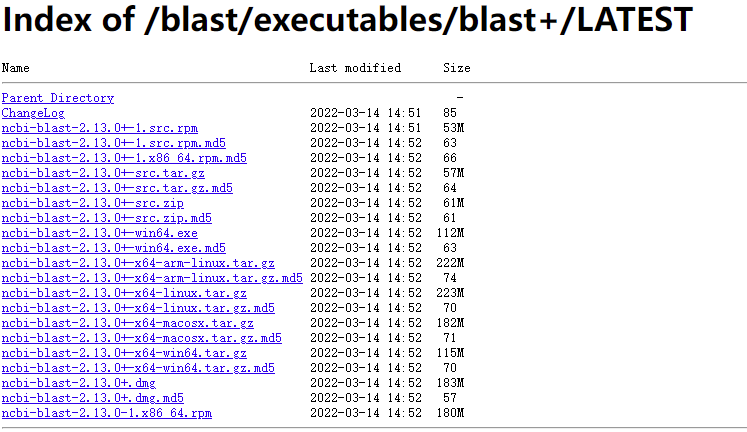
在ncbi官网下载压缩包:https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/:
根据操作系统选择合适的版本,我使用的服务器版本是64位linux操作系统,则选择ncbi-blast-2.13.0+-x64-linux.tar.gz。可以在本地下载,解压缩后上传到服务器上,也可以直接在服务器上下载解压缩。将文件重命名为blast,重命名是为更方便地添加PATH变量,也方便后面地操作,但并不是一定要重命名。
这时进入blast/bin,你可以看到你需要的程序都在里面:blastn,blastp,psi-blast。注意查看以下这些可执行程序的权限,建议都改为777,否则可能因为权限原因之后的命令无法执行。
接下来,把blast路径添加到PATH变量中切换为root用户,执行vim /etc/profile,在最末尾添加:
export PATH=/home/cmq/blast/bin:$PATHPATH的值需要根据实际blast的存放位置做出修改。也可以在你所在的用户目录下的.bashrc文件中修改,添加相同的语句,运行source ~/.bashrc即可。
判断是否安装成功,运行语句:
blastn -version成功显示版本号就代表安装成功。
二、建立本地数据库
根据自己的需求下载合适的数据库,我这里用到的是uniref50.fasta,创建文件夹uniref50-db,并将uniref50.fasta移动到该目录下,并进入该目录:
mkdir uniref50-db
cp uniref50.fasta uniref50-db
cd uniref50-db进入目录后,利用makeblastdb命令建立本地蛋白质序列数据库:
makeblastdb -in uniref50.fasta -dbtype prot -out unirefdb- -in:建立本地数据库需要的fasta文件
- -dbtype:数据库类型,核苷酸的为nucl,蛋白质的为prot
- -out:数据库名称
这是主要的三个参数,还有其他参数可以自行百度。makeblastdb命令比较耗时,可以利用nohup在后台保持运行状态,大约需要1000-2000秒。创建成功后,uniref50-db目录下会出现许多文件:
(nohup.out是nohup命令自动生成的文件)
三、简单的测试
数据库搭建好后,可以利用blastp命令在数据库中找同源序列。
blastp -query demo.fasta -db uniref50-db/unirefdb -out demo.txt有一点很奇怪,uniref50-db目录下没有unirefdb的文件,都是unirefdb带后缀名,但是这样确实能运行。运行成功后,会生成一个txt文件。